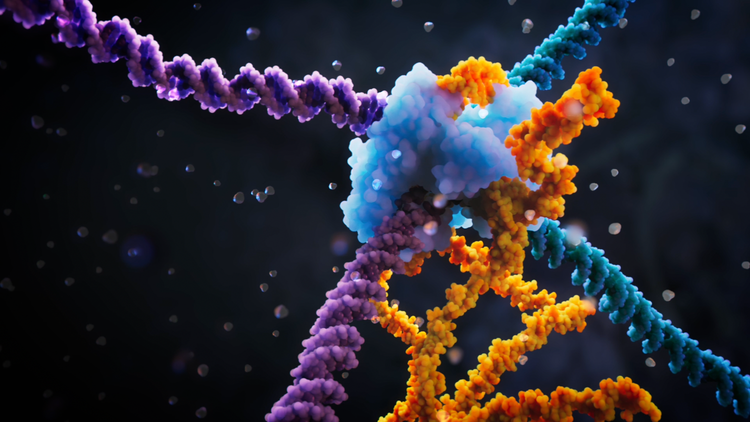
Punktgenaue Veränderungen in Genen ermöglicht die Genschere Crispr. Sie erlaubt rasche Anpassungen von Nutzpflanzen an trockenere, wärmere Verhältnisse und trägt medizinische Hoffnungen: Ende 2023 wurde erstmals eine Gentherapie für Blutkrankheiten wie Sichelzellanämie zugelassen, bei der das revolutionäre Instrument zum Einsatz kommt.
Künftig hat der Baukasten der Molekularbiologie ein neues Werkzeug. Dabei handelt es sich um sogenannte Brücken-RNA: Programmierbare Enzyme können so mit einem einzigen Schritt größere Abschnitte eines Genoms ändern, Sequenzen einfügen oder löschen. Wie das funktioniert, erklären zwei Studien eines US-amerikanisch-japanischen Forschungsteams, die nun im Fachjournal Nature erschienen sind.

Detektivgeschichte
Der Mechanismus "geht weit über programmierbare Genscheren wie Crispr hinaus", heißt es in einer Aussendung des Arc Institute im kalifornischen Palo Alto, dessen Forscher Patrick Hsu leitend an der Entwicklung beteiligt war. Im Vergleich zu bisherigen Ansätzen könnten großangelegte Änderungen des Genoms effizienter und präziser durchgeführt werden. Das Erbgut wird nicht an einer ausgewählten, von RNA angesteuerten Stelle geschnitten und anschließend repariert, sondern neu kombiniert.
Dabei handelt es sich um eine clevere Kombination verschiedener Mechanismen, die auch natürlicherweise auf das Genom einwirken. "Das war eine bemerkenswerte Detektivgeschichte", schrieb Hsu bereits Anfang des Jahres auf X, nachdem er die Methode auf einem Symposium vorgestellt hatte. Sie beginnt mit Transposons, die man auch als "springende Gene" bezeichnet. Wie der Name verrät, ändern diese DNA-Segmente quasi selbstständig ihren Platz im Genom. Die passenden Enzyme heißen Transposasen. Wenn man sie so programmieren kann, dass sie an einem gewünschten Abschnitt eingreifen, lässt sich das für die Veränderung längerer DNA-Abschnitte nutzen – und genau das ist der Gruppe von Patrick Hsu gelungen.
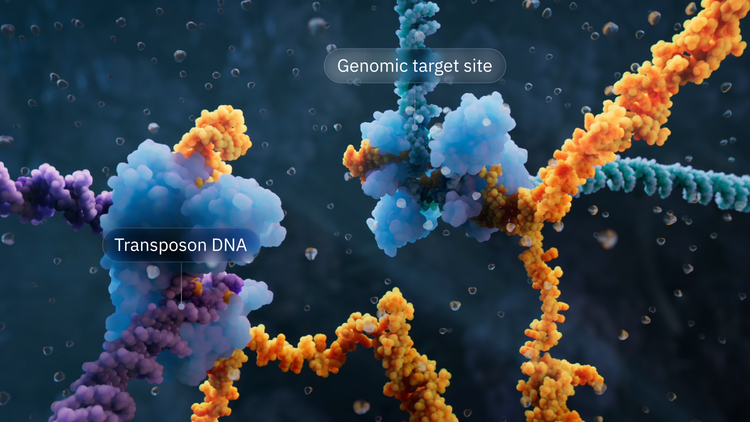
Die Gruppe fand eine bestimmte Variante dieser Transposons, die nicht nur codierende Abschnitte enthält (deren Information sich in Proteine übersetzen lässt), sondern auch erstaunlich lange nichtcodierende Teile. Von solchen Segmenten glaubte man lange Zeit, sie seien unnütz.
"Universeller Adapter"
In diesen Teilen der springenden Gene, die, wie für DNA üblich, normalerweise doppelsträngig sind, fanden Hsu und sein Team allerdings versteckte RNA – die Schwester der DNA, die nur aus einem Strang besteht. Auch sie besteht aus einem nichtcodierenden Buchstabensalat. Durch ihre zwei Schlaufen kann sie aber auch als Brücke verwendet werden, die beim Verändern des Genoms die Ziel-DNA mit der Spender-DNA verbindet.
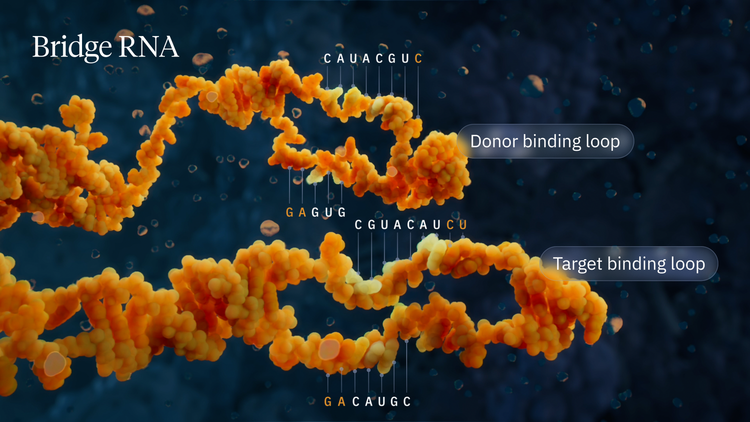
Mithilfe dieser Brücken-RNA entwickelten die Teams von Hsu, der auch an der University of California in Berkeley forscht, Hiroshi Nishimasu von der Universität Tokio und Silvana Konermann von der Universität Stanford programmierbare Enzyme. Sowohl die Stelle, die an das Ziel andockt, als auch die hinzuzufügende Sequenz können voneinander unabhängig angepasst werden. "Das ist so, als ob die Brücken-RNA ein universeller Stromadapter wäre", sagt Autor Nick Perry vom Arc Institute.
Neues Kapitel der Genomveränderung
Laut Erstautor Matthew Durrant löst diese Brückenrekombination "einige der grundlegendsten Probleme, mit denen andere Methoden der Genomeditierung konfrontiert sind". Dass man nun "zwei beliebige DNA-Moleküle programmgesteuert neu anordnen kann, öffnet die Tür für einen Durchbruch beim Genomdesign". Auch die beiden Experten Connor Tou (Massachusetts Institute of Technology) und Benjamin Kleinstiver (Harvard Medical School), die nicht an der Forschung beteiligt waren, schreiben in einer begleitenden Einschätzung, dass dies ein "neues Kapitel für Genomeditierung" sei.
Getestet wurden damit bisher nur Veränderungen in Bakterienzellen. Im E.-coli-Bakterium betrug die Effizienz, ein Gen per Brücken-RNA in das Genom einzubauen, bislang mehr als 60 Prozent. Der angepeilte Zielort in der DNA wurde mit einer Spezifität von mehr als 94 Prozent erreicht. Wie gut das Ganze etwa in Säugetierzellen funktioniert, muss erst noch ermittelt werden. Doch die Methode macht auf Fachleute bisher einen äußerst vielversprechenden Eindruck. (Julia Sica, 26.6.2024)