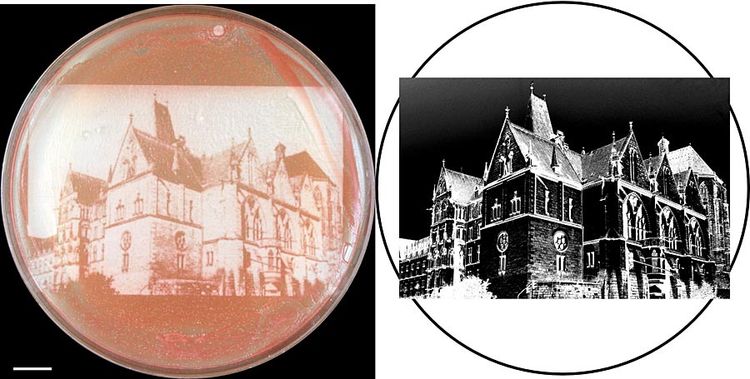
Das Foto der Alten Universität in Marburg wurde mit Hilfe des psd-Moduls in erstaunlich hoher Auflösung auf einem Rasen von Hefezellen reproduziert.
Deutsche Wissenschafter haben ein Werkzeug entwickelt, mit dem sie die Stabilität einzelner Proteine in einer Zelle mit Licht steuern können. Die Forscher koppelten eine selbst entworfene Gen-Sequenz mit einem Lichtsensor der Ackerschmalwand. Dieses Modul lässt sich an beliebige Proteine "montieren", die dann per Lichtsignal zum Abbau freigegeben werden. Hängt man das Modul etwa an ein Protein, das bei der Zerlegung einen Farbstoff entstehen lässt, dann kann man mit lebenden Zellen sogar fotografieren, wie die Forscher von der Philipps-Universität Marburg unter Beweis stellten.
Die Manipulation zellulärer Vorgänge durch Licht bestimmter Wellenlängen hat sich in den letzten Jahren zu einer biotechnologischen Trenddisziplin entwickelt. Doch bislang wurden in dieser als Optogenetik bezeichneten Fachrichtung vor allem Techniken zur Kontrolle der Proteinbiosynthese entwickelt. Der gegenteilige Prozess, der lichtgesteuerte Abbau von Proteinen, ist zwar im Pflanzen- und Tierreich gut bekannt, hat aber noch nicht Einzug in die Biotechnologie gehalten.
Der nun von den Marburger Wissenschaftern um Christof Taxis und Lars-Oliver Essen verfolgte Ansatz ist weitaus vielseitiger und einfacher als der natürliche: Hier wird das ausgesuchte Protein mit einem besonderen Marker verknüpft, um es für den Abbau in der als Proteasom bezeichneten zelleigenen Abbaumaschinerie zu markieren. Das Konstrukt besteht aus einem Lichtsensor der Ackerschmalwand (Arabidopsis thaliana) und einer von den Wissenschaftern entworfenen Sequenz, die den Abbau herbeiführt. Koppelt man dieses sogenannte psd-Modul (photosensitive degron) an ein Zielprotein und setzt die Zelle blauem Licht aus, verändert der Lichtsensor seine dreidimensionale Struktur und macht die Erkennungssequenz so für das Proteasom zugänglich. Dies beginnt den Abbau des Konstruktes samt Zielprotein.
Fotos mit Bäckerhefe
Da der Abbau über das Proteasom in allen höheren Lebewesen gleich abläuft, sollte das neue Modul auch in Pflanzen und Wirbeltieren einsetzbar sein; die Funktionsweise des photosensitiven Degrons haben die Forscher eindrücklich in Experimenten mit der Bäckerhefe (Saccharomyces cerevisiae) gezeigt. So ist es ihnen etwa gelungen, ein Foto der Alten Universität in Marburg auf einem Rasen von Hefezellen zu reproduzieren.
Dazu koppelten sie ihr psd-Modul an ein Protein, dessen Abbau zu einer Anreicherung von rotem Farbstoff in den Zellen führt. Über diese Zellschicht legten sie dann ein Schwarz-Weiss-Negativ des Fotos und beleuchteten sie mit blauem Licht. In der Folge entwickelte sich ein rötlich-beiges Abbild der Alten Universität in beeindruckend hoher Auflösung auf dem Hefe-Rasen. In einem anderen Versuch fusionierten die Forscher ihr Konstrukt mit einem Protein, das für das Wachstum von Hefezellen essenziell ist. In der Kulturschale wuchsen die Zellen daraufhin nur im Schatten einer sternförmigen Maske, der sie vor dem blauen Licht – und damit vor dem Licht-induzierten Abbau des Wachstumsfaktors – schützte. (red, derStandard.at, 20.04.2013)