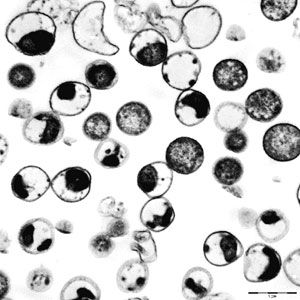
Die sogenannten Elementarkörperchen sind eine eigene Zellform, die für die Infektion neuer Wirtszellen zuständig ist.
Die Natur hat es der Wissenschaft nicht leichtgemacht: Die DNA eines Lebewesens zu entschlüsseln, das mag heute zwar schon fast zum Standardrepertoire molekularbiologischer Labore gehören, aber das scheinbar wilde biochemische Chaos im Inneren einer lebendigen Zelle zu entschlüsseln - das steht noch auf einem ganz anderen Blatt. Eine fast unüberschaubare Vielfalt an unterschiedlichen Proteinen spielt in diesem Gemisch die Hauptrolle. Fachleute bezeichnen es als Proteom. Dieser Begriff wurde erst Anfang der Neunziger geprägt.
Das Proteom ist das praktisch funktionelle Gegenstück zum Genom. Bevor man die Wirkung der verschiedenen Proteinmoleküle - vor allem untereinander - untersuchen kann, muss man sie zuerst identifizieren. Hierauf haben sich die Experten des Christian-Doppler-Labors für Proteomik in Wien spezialisiert - mit Erfolg. "Vor zehn, 15 Jahren waren die Methoden und Ansätze noch ein bisserl mühsamer", sagt Laborleiter Gustav Ammerer. Heute verfügt man über moderne Techniken der Massenspektrometrie. Sie ermöglichen die Erkennung zahlreicher unterschiedlicher Proteinsorten in kurzer Zeit. An den Methoden wird ständig weitergearbeitet. "Die Vision ist, dass man alle Proteine und deren Modifikationen in einem einzelnen Messvorgang feststellen kann", sagt Ammerer.
Eine der aktuellsten wissenschaftlichen Entdeckungen, die mit Beteiligung des CD-Labors für Proteomik zustande kam, betrifft die komplexe Lebensweise von Protochlamydia amoebophila, ein naher Verwandter der als Krankheitserreger gefürchteten Chlamydien. Keime der Art Chlamydia pneumoniae lösen beim Menschen Lungenentzündungen aus, Chlamydia trachomatis dagegen Geschlechtskrankheiten sowie Trachoma, ein schweres Augenleiden. Diesen Bakterien ist nicht leicht beizukommen. Sie verbringen einen Großteil ihres Lebenszyklus als Parasiten versteckt in Körperzellen. Und vermehren sich sogar darin.
Diese Eigenschaft teilt Chlamydia mit allen anderen bekannten Arten der Chlamydiae-Gruppe - darunter auch P. amoebophila. Dessen Proteom wurde nun erstmalig von einem Expertenteam unter Leitung des Mikrobiologen Matthias Horn von der Universität Wien durchleuchtet. P. amoebophila lebt normalerweise in Amöben und ist den humanpathogenen Chlamydien genetisch sehr ähnlich.
Der besondere, zweiphasige Lebenszyklus der Chlamydiae fasziniert Forscher schon seit längerem. In den Wirtszellen findet man in erster Linie die sogenannten Retikularkörper. Sie bewohnen einen durch eine Membran abgegrenzten Zellbereich. Die winzigen Untermieter bauen quasi ihr eigenes Kämmerlein. Die Retikularkörper vermehren sich über Zweiteilung. Danach bildet sich eine ganz andere Zellform, die Elementarkörperchen (EK), die aus der Wirtszelle freigesetzt werden und draußen neue Opfer befallen. Eine Art Generationswechsel. "Das hat man sonst bei Bakterien nicht", betont Matthias Horn.
Eiweißausstattung
Horns Team widmete sich in seiner P.-amoebophila-Studie vor allem der Eiweißausstattung der Elementarkörperchen. Das Projekt wurde finanziell unter anderem vom Wissenschaftsfonds FWF und der Österreichischen Akademie der Wissenschaften unterstützt. "Die Proteomik ist ganz wichtig, um zu bestimmen, welche im Genom kodierten Proteine wann welche Rolle spielen", erklärt Horn. Mit anderen Worten: Wer wissen will, wie die zelluläre Maschinerie im realen Leben funktioniert, muss auf die Proteine schauen, und nicht so sehr auf die Gene.
Bisher glaubten Fachleute, die Elementarkörperchen der Chlamydien seien so etwas wie physiologisch in sich ruhende Samenkörner. Offenbar ein Irrtum. Matthias Horn und seine Kollegen stellten begeistert fest, dass in diesem Zellstadium von P. amoebophila 472 verschiedene Proteine mit zahlreichen Funktionen sehr häufig vorkommen. Horn: "Die Elementarkörperchen sind wohl nicht so inaktiv, wie man das immer dachte."
Zur EK-Proteinausstattung gehören allerdings nicht nur Enzyme mit Funktionen in der Energieversorgung und der Synthese neuer Proteine, sondern auch Eiweißmoleküle, die bei der aktiven Infektion von neuen Wirtszellen zentrale Rollen spielen dürften. Eine komplette Übersicht der Untersuchungsergebnisse wurde neulich im internationalen Fachblatt Proteomics veröffentlicht (Bd. 11, S. 1868).
Mittel gegen uralte Bakterien
Da der Stoffwechsel von P. amoebophila sehr gut mit der Physiologie von krankheitserregenden Chlamydien vergleichbar ist, dürften sich hier zukünftig interessante medizinische Perspektiven eröffnen, meint die Wiener Biologin Barbara Sixt, Erstautorin der neuen Studie. "Man könnte so mögliche Ziele für Medikamente identifizieren." Wer die Stoffwechselprozesse und die Infektionsmechanismen der Keime im Detail kennt, kann auch leichter neue Präparate entwickeln, um genau diese Vorgänge zu blockieren.
Diese vergleichenden Untersuchungen ermöglichen auch tiefere Einblicke in die Evolution der Chlamydiae. Die Vorfahren dieser Bakterien haben vor mindestens 700 Millionen Jahren Methoden erfunden, um in Einzellern wie Amöben überleben zu können, erläutert Matthias Horn. Auf Dauer war das offenbar ein Erfolgsrezept. Humanpathogene Chlamydien sind dagegen erst viel später entstanden. Den Proteomikergebnissen nach verfügen die urtümlicheren Protochlamydia gleichwohl über mehr aktive Proteine als ihre an Menschen angepassten Verwandten. Dieser Vielfalt ist den Krankheitserregern im Laufe ihrer Entwicklung verlorengegangen. Vermutlich wurde sie einfach nicht mehr benötigt. (Kurt de Swaaf, DER STANDARD, Print-Ausgabe, 25. Mai 2011)