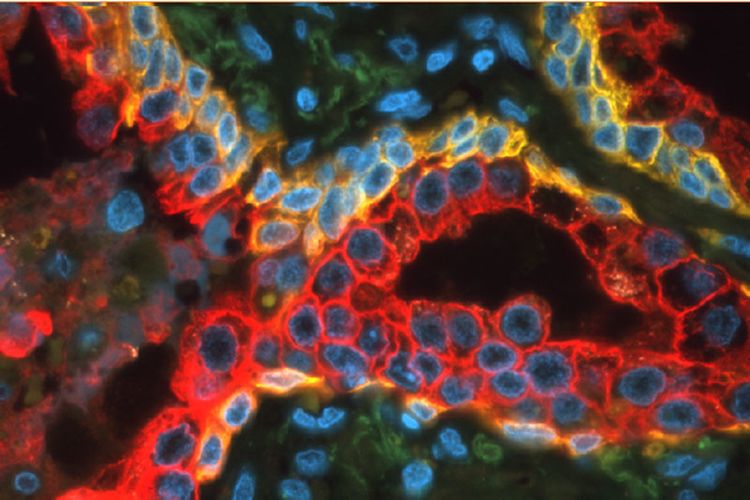
Tumorgewebe mit Antigenen, die mit verschiedenen Farben markiert sind, damit sie dann elektronisch exakt quantifiziert werden können.
Wissenschafter der Veterinärmedizinischen Universität Wien, des Ludwig Boltzmann Instituts für Krebsforschung und der MedUni Wien haben gemeinsam mit dem Wiener Unternehmen "Tissuegnostics" an Gewebeproben von Leberkarzinompatienten ein neues System zur exakten Quantifizierung im Tumorgewebe vorkommender Proteine und von DNA getestet. Das könnte die Charakterisierung von Tumoren vereinfachen.
"Wir haben dieses Verfahren der Vergangenheit bereits seit in der Forschung verwendet. Jetzt haben wir Gewebeschnitte von 30 Patienten mit Leberzellkarzinomen analysiert", sagt Lukas Kenner von der Abteilung für Labortierpathologie der VetMed-Uni. Die entsprechende Studienarbeit ist jetzt in "Plos One" erschienen.
Proteine identifizieren
Bei der Entwicklung geht es um die Schaffung eines Systems, mit dem man möglichst einfach, genau, objektiv und nachvollziehbar die für die jeweils untersuchten (Krebs-)Zellen charakteristischen Proteine identifizieren und quantitativ genau messen kann. "Das ist für die Diagnose, die Feststellung der Erkrankungsart und die Behandlung mit Arzneimitteln der zielgerichteten Therapie wichtig", sagt Kenner.
Bisher erfolgten solche Untersuchungungen durch die Pathologen an Gewebeschnitten, indem man beispielsweise Antikörper zur Markierung von Proteinen hinzufügte und dann im Mikroskop zwischen "negativ" für ein untersuchtes Proteinmerkmal, "+" (positiv), "++" oder gar "+++" (hochgradig positiv) unterschied. Der Wiener Wissenschafter: "Allerdings waren sich in ihren jeweiligen Einschätzungen zwei unabhängig von einandere begutachtende Pathologen nur bei jeder dritten Diagnose einig."
Neues System
Mit dem neuen System aus hochsensibler Digitalfotografie und einem speziell entwickelten Computerprogramm kann jetzt eine echte Quantifizierung der untersuchten Proteine, etwa auch von typischen Rezeptoren an der Zelloberfläche, auf objektive Weise erreicht werden.
Sie bleibt auch für die Zukunft gespeichert - das System ist somit feiner als die bisherige Einteilung nach Kategorien. "Die neue Software wird auch dazu beitragen, besser abwägen zu können, wo eine teure Therapie gerechtfertigt ist, aber auch, in welchem Falle sie nicht nötig ist und dem Betroffenen erspart werden kann", meinte der Wissenschafter.
Möglicher "Riesenfortschritt"
Die Wissenschafter etablierten das System zunächst für die Messung der Proteine Stat5 und JunB, die beide eine Rolle bei Krebserkrankungen haben. Dies erfolgte am Modell von Labormäusen, bei denen man künstlich die Gene für diese Eiweißstoffe ganz oder Hälfte ausschalten kann. Als das Verfahren entsprechende Ergebnisse erbrachte, gingen die Wissenschafter den nächsten Schritt: Sie bestimmten diese Proteine an Feinschnitten von in Paraffin eingelegten Gewebeproben von Leberkarzinompatienten.
Kenner bezeichnete das System als einen möglichen "Riesenfortschritt". Natürlich werde es die Einschätzung der Pathologen nicht ersetzen. "Die neu entwickelte Software bietet aber erstmals die Möglichkeit, die sogenannte 'Inter-Observer-Variabilität', also die Unterschiede im Urteil der beobachtenden Personen, auszuschalten." (APA, derStandard.at, 14.7.2014)